Introduction
L’arbre phylogénétique est une représentation graphique des relations évolutives entre les espèces‚ décrivant leur histoire évolutive et leurs liens de parenté.
Définition de l’arbre phylogénétique
L’arbre phylogénétique est une représentation graphique qui décrit les relations évolutives entre les espèces‚ montrant comment elles sont liées les unes aux autres par des ancêtres communs.
Cette représentation permet d’étudier l’évolution des espèces au fil du temps‚ en reconstruisant leur histoire évolutive à partir de caractères morphologiques et moléculaires.
L’arbre phylogénétique est donc un outil essentiel en biologie évolutive‚ car il permet de comprendre les processus évolutifs qui ont mené à la diversité des espèces actuelles.
La construction d’un arbre phylogénétique repose sur l’analyse de différentes données‚ telles que les caractères morphologiques‚ les séquences d’ADN ou les propriétés biochimiques‚ pour identifier les relations évolutives entre les espèces.
Caractéristiques de l’arbre phylogénétique
L’arbre phylogénétique présente des caractéristiques spécifiques‚ telles que la représentation des relations évolutives‚ la classification biologique‚ l’identification des ancêtres communs et la visualisation de l’évolution des espèces.
Représentation des relations évolutives
L’arbre phylogénétique représente les relations évolutives entre les espèces à travers une structure hiérarchique‚ où chaque nœud correspond à un ancêtre commun et chaque branche symbolise la divergence évolutive.
Cette représentation permet de visualiser les liens de parenté entre les espèces‚ ainsi que les processus évolutifs tels que la spéciation‚ la radiation adaptative et l’extinction.
Les relations évolutives sont déduites à partir d’analyses de données génétiques‚ morphologiques ou moléculaires‚ qui permettent de reconstruire l’histoire évolutive des espèces.
L’arbre phylogénétique est ainsi un outil puissant pour comprendre les mécanismes évolutifs et les processus qui ont modelé la biodiversité actuelle.
Éléments clés de l’arbre phylogénétique
L’arbre phylogénétique est composé de plusieurs éléments clés qui permettent de comprendre les relations évolutives entre les espèces.
- Nœuds ⁚ représentent les ancêtres communs et les points de divergence évolutive.
- Branche ⁚ symbolise la divergence évolutive et la distance génétique entre les espèces.
- Longueur de branche ⁚ représente le temps écoulé entre deux événements évolutifs.
- Rotation de nœuds ⁚ permet de modifier l’orientation des branches pour améliorer la lisibilité de l’arbre.
Ces éléments clés permettent de décrypter l’histoire évolutive des espèces et de comprendre les mécanismes qui ont modelé la biodiversité actuelle.
Types d’arbres phylogénétiques
Les arbres phylogénétiques se divisent en deux grandes catégories ⁚ les arbres phylogénétiques morphologiques et les arbres phylogénétiques moléculaires.
Arbres phylogénétiques morphologiques
Ces arbres sont basés sur l’étude des caractères morphologiques‚ tels que la forme et la structure des organes‚ pour établir les relations évolutives entre les espèces.
Ils utilisent des données issues de l’anatomie comparée‚ de la paléontologie et de la biologie du développement pour reconstruire l’histoire évolutive des organismes.
Les caractères morphologiques sont souvent utilisés pour définir les groupes monophylétiques‚ c’est-à-dire les groupes d’organismes qui partagent un ancêtre commun.
Ces arbres sont particulièrement utiles pour l’étude des fossiles et des groupes d’organismes qui ne possèdent pas de séquences d’ADN ou d’ARN conservées.
Toutefois‚ les arbres phylogénétiques morphologiques peuvent être affectés par les limitations de la fossilisation et de la préservation des caractères morphologiques.
Arbres phylogénétiques moléculaires
Ces arbres sont basés sur l’analyse des séquences d’ADN ou d’ARN pour établir les relations évolutives entre les espèces.
Ils utilisent les données génétiques pour reconstruire l’histoire évolutive des organismes‚ en comparant les séquences de gènes ou de régions génomiques spécifiques.
Les arbres phylogénétiques moléculaires permettent de déterminer les distances évolutives entre les espèces‚ en mesurant le nombre de mutations accumulées dans les séquences d’ADN ou d’ARN.
Ces arbres sont particulièrement utiles pour l’étude des relations évolutives entre les espèces qui ont des fossiles rares ou absents‚ ou pour lesquelles les caractères morphologiques sont peu informatifs.
Les techniques de phylogénétique moléculaire‚ telles que la PCR et le séquençage à haut débit‚ ont révolutionné la reconstruction des arbres phylogénétiques.
Méthodes d’inférence phylogénétique
Les méthodes d’inférence phylogénétique permettent de reconstruire l’arbre phylogénétique à partir de données morphologiques‚ moléculaires ou combinées‚ en utilisant des algorithmes et des modèles statistiques complexes.
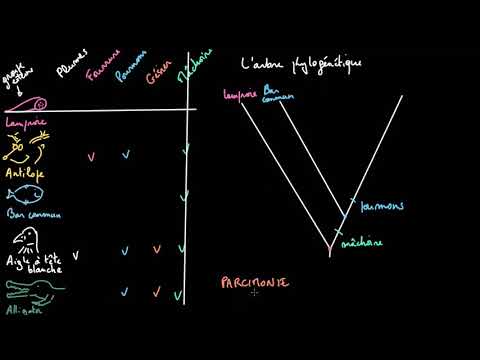
Analyse de parcimonie
L’analyse de parcimonie est une méthode d’inférence phylogénétique qui vise à trouver l’arbre phylogénétique le plus parcimonieux‚ c’est-à-dire celui qui nécessite le minimum de changements évolutifs pour expliquer les données observées.
Cette méthode repose sur l’idée que les événements évolutifs sont rares et que les caractères ancestraux sont plus probables que les caractères dérivés.
L’analyse de parcimonie utilise des algorithmes spécifiques pour explorer l’espace des arbres possibles et sélectionner celui qui minimise le nombre de changements évolutifs.
Cette approche est particulièrement utile pour l’analyse de grandes quantités de données et permet de reconstruire des arbres phylogénétiques robustes et bien résolus.
L’analyse de parcimonie est souvent associée à d’autres méthodes‚ comme la rotation de nœuds et la longueur de branche‚ pour améliorer la résolution de l’arbre phylogénétique.
Rotation de nœuds et longueur de branche
La rotation de nœuds et la longueur de branche sont deux concepts clés en analyse phylogénétique qui permettent de représenter les relations évolutives entre les espèces de manière plus précise.
La rotation de nœuds consiste à modifier la position des nœuds internes de l’arbre phylogénétique pour révéler les relations évolutives cachées.
La longueur de branche‚ quant à elle‚ représente la distance évolutionnaire entre deux nœuds consécutifs de l’arbre.
En combinant ces deux concepts‚ il est possible de reconstruire des arbres phylogénétiques plus résolus et plus précis‚ qui tiennent compte des événements évolutifs complexes qui ont conduit à la diversification des espèces.
Ces approches sont particulièrement utiles en cladistique et en phylogénétique moléculaire‚ où la précision de la reconstruction de l’arbre phylogénétique est cruciale.
Exemples d’arbres phylogénétiques
L’étude de la phylogénie a permis de reconstruire des arbres phylogénétiques pour de nombreux groupes d’organismes‚ tels que les vertébrés‚ les plantes à fleurs et les bactéries.
Arbre phylogénétique des vertébrés
L’arbre phylogénétique des vertébrés est un exemple classique de la reconstruction des relations évolutives entre les espèces. Cette analyse phylogénétique a permis de déterminer les liens de parenté entre les différentes classes de vertébrés‚ telles que les poissons‚ les amphibiens‚ les reptiles‚ les oiseaux et les mammifères.
Cet arbre phylogénétique montre que les vertébrés partagent un ancêtre commun qui a vécu il y a environ 500 millions d’années. Les poissons sont apparus en premier‚ suivis par les amphibiens‚ les reptiles‚ les oiseaux et enfin les mammifères.
Cette reconstruction phylogénétique est basée sur l’analyse de caractéristiques morphologiques et moléculaires‚ telles que la structure du squelette‚ la forme des dents et les séquences d’ADN. L’arbre phylogénétique des vertébrés est un outil précieux pour comprendre l’évolution des espèces et leurs relations de parenté.
Arbre phylogénétique des plantes à fleurs
L’arbre phylogénétique des plantes à fleurs‚ également connu sous le nom d’angiospermes‚ est un exemple remarquable de la diversité évolutive des espèces végétales.
Cette analyse phylogénétique a permis de reconstruire l’histoire évolutive des plantes à fleurs‚ qui ont émergé il y a environ 130 millions d’années. L’arbre phylogénétique des plantes à fleurs montre que ces dernières partagent un ancêtre commun avec les gymnospermes‚ telles que les conifères et les cycadées.
L’analyse des caractéristiques morphologiques et moléculaires‚ telles que la structure des fleurs et les séquences d’ADN‚ a permis de déterminer les liens de parenté entre les différentes familles de plantes à fleurs‚ telles que les rosidées‚ les asteridées et les lamiidées.
L’arbre phylogénétique est un outil puissant pour comprendre les relations évolutives entre les espèces et reconstruire l’histoire de la vie sur Terre.
Grâce à l’analyse des caractéristiques morphologiques et moléculaires‚ ainsi qu’à l’utilisation de méthodes d’inférence phylogénétique telles que l’analyse de parcimonie et la rotation de nœuds‚ les scientifiques peuvent reconstruire les arbres phylogénétiques qui décrivent les liens de parenté entre les espèces.
Les exemples d’arbres phylogénétiques‚ tels que ceux des vertébrés et des plantes à fleurs‚ montrent la richesse et la complexité de l’évolution des espèces. En fin de compte‚ l’étude de l’arbre phylogénétique permet de mieux comprendre la biodiversité et de préserver les espèces menacées.
Je tiens à féliciter l
J
Je suis satisfait de voir que l
Je suis impressionné par la façon dont l
Je recommande cet article à tous ceux qui s
Je trouve que l